聯系電話
- 聯系人:
- 曹女士
- 電話:
- 400-6111-883
- 手機:
- 售后:
- 4006-111-883
- 傳真:
- 86-21-34615995
- 地址:
- 上海市浦東新區天雄路166弄1號3樓
- 網址:
- www.yeasen.com
掃一掃訪問手機商鋪
phi29 DNA聚合酶是一種從Bacillus subtilis噬菌體phi29中克隆出來的嗜溫DNA聚合酶,具有很強的鏈置換活性、持續合成能力和高的保真度,是滾環擴增(RCA)和多重鏈置換擴增(MDA)的用酶。目前,它在DNA酶法合成、全基因組擴增、納米孔測序儀開發等領域發揮著重要作用,應用潛力巨大。接下來,小編將從phi29 DNA聚合酶的結構、活性、應用等方面進行展開介紹。
phi29 DNA聚合酶結構及活性
Phi29 DNA聚合酶是一種分子量約為66 kda的單體酶,該酶由多個關鍵結構域構成,包括核酸外切酶結構域、Thumb結構域、Palm結構域,以及兩個重要的TPR結構域,具有5'-3'聚合酶活性、3'-5'核酸外切酶活性和鏈置換活性,在生物學研究及應用中具有廣泛的應用前景。
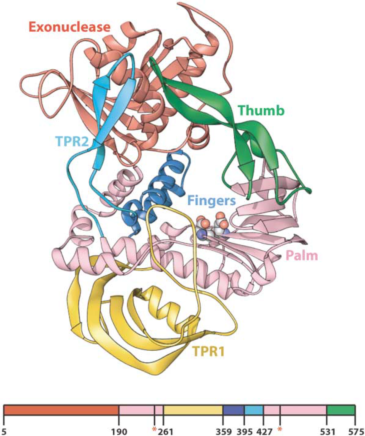
圖1. phi29 DNA 聚合酶結構[1]
3’-5’核酸外切酶活性(校正活性)
Phi29 DNA 聚合酶有3’-5’核酸外切酶結構域,具備高保真(校正)活性,其保真度是Taq酶的1000倍,高于目前絕大多數高保真酶的保真度,可保證擴增反應的高保真性。
聚合活性
phi29 DNA聚合酶的聚合活性源于其Palm、Thumb和Fingers結構域組合,共同構建了一個高效的聚合活性區域。這種特殊的結構賦予了它5'-3'聚合酶活性,使其對單鏈DNA或單鏈RNA展現出底物親和力和強大的持續合成能力。在單次聚合反應中,phi29 DNA聚合酶能夠實現超過70kb的連續聚合延伸。
鏈置換活性
phi29 DNA聚合酶的兩個亞結構域——TPR1和TPR2的存在,使得phi29 DNA聚合酶展現出了強大的鏈置換活性,能夠有效地解鏈和復制DNA的復雜結構。因此,在體外環境下,phi29 DNA聚合酶能夠執行不依賴于熱循環的等溫DNA聚合反應,展現出其DNA復制能力。
phi29 DNA聚合酶相關的主流DNA擴增技術
相較于常規DNA聚合酶,phi29 DNA聚合酶展現出了持續合成能力和鏈置換活性,其保真度也遠超常規聚合酶。這些顯著的優勢使得phi29 DNA聚合酶在多種DNA擴增技術中得到了廣泛應用,如多重置換擴增(MDA)和滾環擴增(RCA)等。接下來,我們將詳細介紹phi29 DNA聚合酶在這些主要技術中的表現和應用前景。
多重置換擴增(MDA)
多重置換擴增(MDA)的核心原理在于利用隨機的六聚體引物與phi29 DNA聚合酶在恒溫條件下協同作用,以實現DNA的高效擴增。這一過程依賴于phi29 DNA聚合酶的持續合成能力、高保真度及鏈置換活性(實現等溫擴增的關鍵)。
在反應過程中,六聚體引物隨機結合至DNA模板之上,隨后phi29 DNA聚合酶在DNA的多個位點同步啟動復制過程。新合成的DNA逐步取代模板的互補鏈,而被置換的互補鏈則能夠作為新的模板繼續參與擴增反應。MDA憑借其能夠從微量樣本中產出大量高質量DNA的能力,已成為當前單細胞全基因組擴增領域最為廣泛應用的技術之一。

圖2. MDA原理圖[2]
滾環擴增(RCA)
滾環擴增(RCA)是一種模擬自然界微生物環狀DNA滾環復制過程的核酸擴增方法。該方法以環狀DNA為模板,借助一個與部分環狀模板互補的短DNA引物,在phi29 DNA聚合酶(具備強持續合成能力、高保真度及鏈置換活性)的催化作用下沿環狀模板延伸,并持續置換先前產生的延伸鏈。最終,這一過程將產生重復的長單鏈DNA產物。通過在RCA基礎上引入與延伸產物退火雜交的另一條或多條引物,還可以形成超分支RCA,實現超指數式擴增。RCA技術以其高靈敏度、良好特異性和易于操作的特性,在SNP檢測、質粒體外酶促生產、DNA測序模板制備等領域具有廣泛的應用潛力。
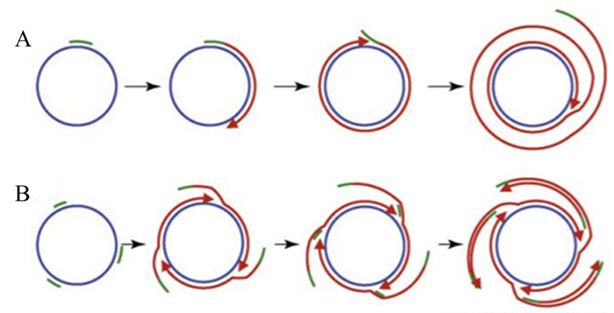
圖3. RCA原理圖[3]
A: 以單引物擴增產生長鏈DNA;B: 以隨機引物擴增產生長鏈串聯DNA
phi29 DNA聚合酶相關的主流應用
DNA酶法合成
質粒DNA,作為mRNA疫苗、DNA疫苗以及細胞基因治療等多個領域的核心“元件",扮演至關重要的角色。傳統的質粒生產方式涉及大腸桿菌發酵和多級放大擴增過程,最終進入純化環節。然而,發酵過程中的不可控風險因素限制了高質量質粒載體的產量,這成為了疫苗產能的關鍵瓶頸。
英國的Touchlight公司創新性地推出了doggybone DNA(dbDNA)載體技術。這項技術了傳統的細菌發酵制備質粒DNA的方法,使體外酶法合成DNA成為可能。
這種創新方法帶來了以下顯著優勢:
速度快,產量高:體外酶促法僅需數周即可生產克級dbDNA(常規質粒生產通常是毫克級),比傳統的細菌發酵法生產質粒DNA快5倍。
優化載體:dbDNA可以編碼更長、更復雜或更不穩定的基因,且無需細菌DNA和抗性篩選,避免生物發酵過程的諸多不可控風險。
dbDNA體外酶法合成:
dbDNA是一種小的、線性的雙鏈共價閉合DNA結構,其形狀類似于“狗骨頭"(doggybone),可以編碼長片段、復雜或不穩定的DNA序列,由3個部分組成:兩端的TEL序列(TelN端粒酶切割后的互補的閉合結構),及中間的目標DNA序列。
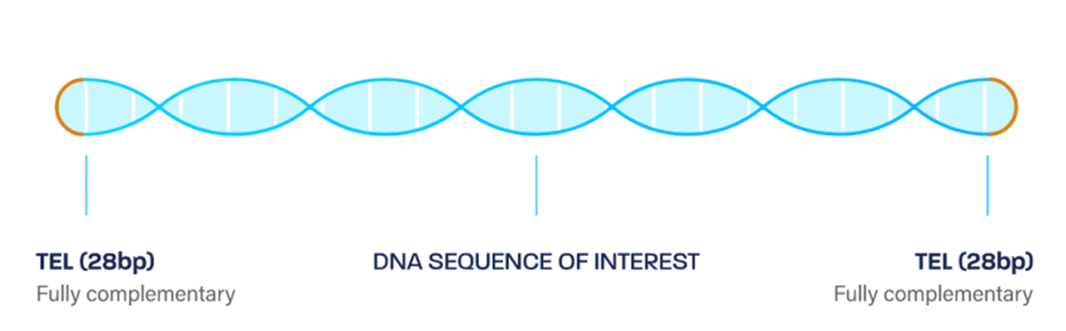
圖4. dbDNA結構(來自Touchlight)
dbDNA體外酶法合成工藝:
模板變性
dbDNA的起始模板為環狀質粒DNA,包含目標基因序列、端粒酶識別序列(telRL)和細菌骨架序列。通過變性處理,將其轉化為兩個單鏈環狀DNA(圖5,AB)。
滾環擴增
利用Phi29 DNA聚合酶,以單鏈環狀DNA為模板,進行滾環擴增,生成具有端粒酶識別序列間隔的長線性雙鏈多聯體DNA(圖5,CDE)。
切割及共價閉合
源自N15噬菌體的端粒酶TelN識別多聯體DNA上的端粒酶識別序列(telRL)發揮切割和連接活性,生成線性、末端共價連接的mini DNA單體(圖5,FG)。
去除細菌骨架DNA
端粒酶處理后的體系中包含細菌骨架序列DNA(backbone DNA)和目標基因DNA(dbDNA)兩部分。細菌骨架DNA其末端具有開放結構,可通過添加限制性內切酶和核酸外切酶降解。最后,對反應體系的成分和降解的核酸片段進行純化,得到僅含有目標基因表達元件的dbDNA。
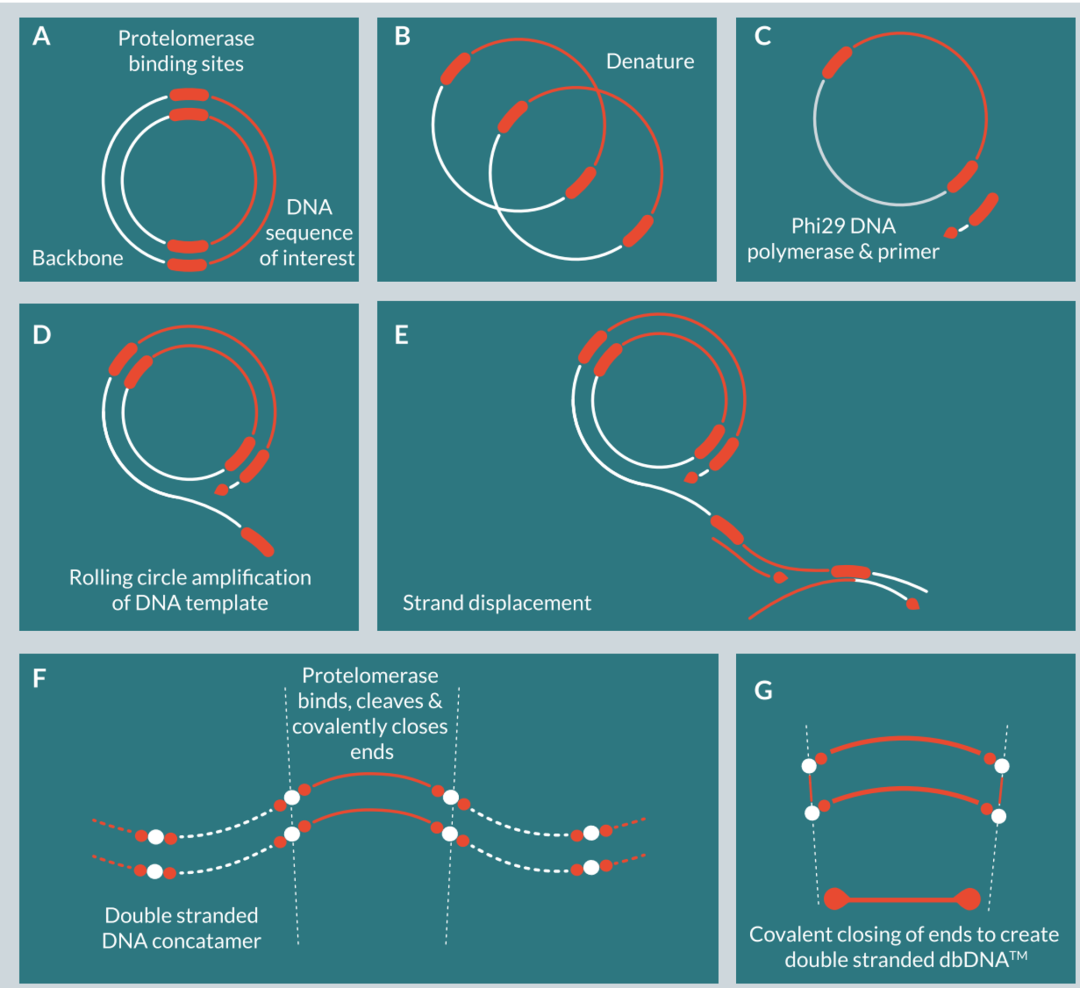
圖5. dbDNA體外酶法合成工藝(來自Touchlight)
這種體外DNA酶法合成技術以其優勢,能夠巧妙地規避生物發酵過程中存在的多種不可預測和難以控制的風險。正因如此,它為眾多新興技術,如mRNA疫苗、DNA疫苗、基因治療以及基因編輯等提供了強有力的支持,并展現出了極為廣闊的應用前景。翌圣生物可提供DNA酶法合成的核心酶原料:phi29 DNA聚合酶(Cat#14404ES)及TelN端粒酶(Cat#4540ES)等,助力DNA酶法合成技術研發生產。
單細胞基因組測序
單細胞測序是研究細胞間差異的一種重要技術,但由于單個細胞的基因組DNA含量較低(pg級別),無法滿足各大測序平臺上機要求,需要通過全基因組擴增技術(WGA)來解決該問題。其中MDA是目前應用廣泛的單細胞全基因組擴增技術。其操作簡單,對實驗儀器的要求低,使用非預變性的模板時也可取得良好的效果,可進一步簡化實驗操作及避免污染的產生,廣泛應用于基因組測序、SNPs 分析等相關領域。
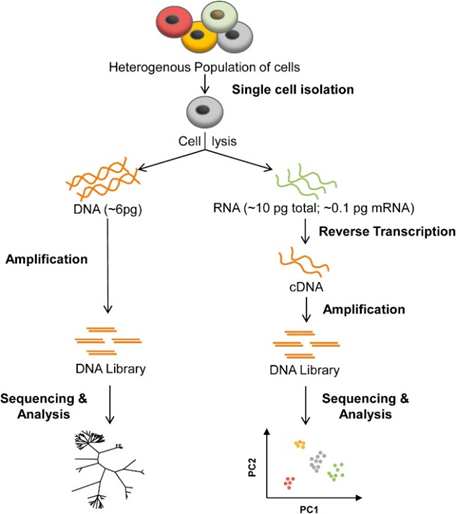
圖6. 單細胞測序流程圖[4]
納米孔測序儀開發
第三代測序技術是指單分子實時測序技術,也叫從頭測序技術,其最大的特點就是單分子測序,測序過程無需進行PCR擴增,實現了對每一條DNA分子的單獨測序。目前已應用于基因組測序、甲基化研究和突變鑒定等多個研究領域。英國牛津納米孔公司所開發的納米孔(Nanopore)測序技術便是三代測序的代表性技術之一。
其優勢主要有:
長讀長:Reads可達Mb;
實時獲得序列信息:最快可在1小時內完成測序流程及數據分析;
裝置便攜:重量輕,體積小,可以隨身攜帶;
設備低成本:測序芯片可清洗再生,重復利用;
直接測序:不需要進行PCR擴增,直接測序原始DNA和RNA,可避免擴增偏好性,保留原始堿基修飾信息,能夠直接檢測胞嘧啶的甲基化狀態。
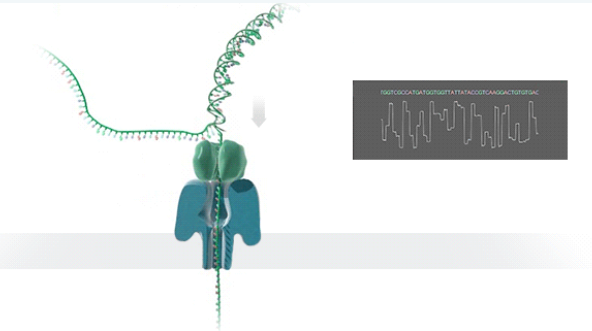
圖7. 納米孔測序原理圖(來自Oxford Nanopore Technologies)
納米孔測序原理:
納米孔測序基于電信號,核心是利用一個納米孔,孔內共價結合特定的分子接頭,將納米孔蛋白固定在電阻膜上后,利用動力蛋白牽引核酸穿過納米孔,當核酸分子通過納米孔時,會引起電荷狀態的變化,進而導致電阻膜上電流發生波動。納米孔直徑通常比DNA分子直徑小一個數量級,保證只有一個DNA分子通過每一個納米孔,ATCG單個堿基的帶電性質不一樣,因此不同堿基通過蛋白納米孔時對電流產生的干擾不同,通過實時監測并解碼這些電流信號便可確定堿基序列,從而實現測序。
納米孔測序關鍵元件的關鍵元件之一是Motor——馬達蛋白。在文庫構建時,需要在接頭上連接一種馬達蛋白,用于將DNA或RNA分子推入納米孔中。常見的Motor包括DNA聚合酶類的Phi29 DNA聚合酶或其變體、解旋酶類的RecD解旋酶、TraI解旋酶、Dda解旋酶等。
目前國內外有多家公司正在布局或已有納米孔測序儀器上市,在儀器開發中,天然的phi29 DNA聚合酶或解旋酶,往往難以滿足高標準的測序要求。為此,翌圣生物不僅可提供商業化分子酶產品(如phi29 DNA聚合酶Cat#14404ES等),還針對在測序儀器開發等應用中可能面臨的市售酶或自然酶性能不足的問題,成立以酶進化技術為驅動、專注于提供酶改造定制化解決方案的子公司-鎂孚泰生物(點擊了解更多)。依托于翌圣生物,鎂孚泰生物現有六大技術平臺:ZymeEditor創新酶進化平臺、多宿主高效表達平臺、發酵工藝開發平臺、純化工藝開發平臺、超潔凈酶生產平臺以及酶分析與質控平臺。基于這六大技術平臺,鎂孚泰生物可提供酶定向改造、新酶開發、酶工藝開發以及GMP級別規模化生產的全套定制解決方案,以滿足酶在體外診斷、生物醫藥、合成生物、醫療美容、醫藥中間體以及測序等領域的應用需求。
若您有酶定制的需求,在線填寫需求表或掃描下方二維碼聯系我們吧!
點擊在線填寫定制服務需求表

翌圣phi29 DNA 聚合酶性能展示
高靈敏度:可對pg級別的DNA模板進行有效擴增
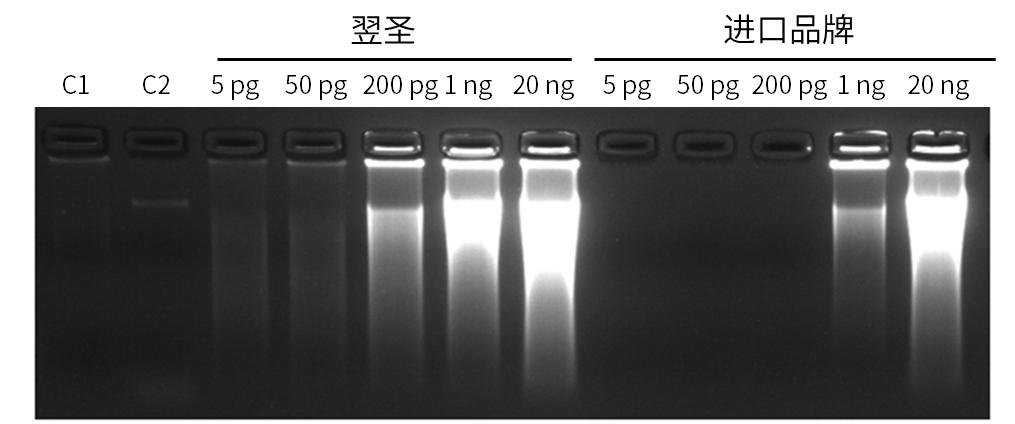
圖9.翌圣及進口品牌phi29 DNA聚合酶不同DNA模板投入量擴增結果。C1:無模板對照,C2:無酶對照。結果顯示翌圣phi29 DNA聚合酶(Cat#14404ES)靈敏度及擴增產量優于進口品牌。
高擴增產量:100ng模板投入,產量可達2 μg/μL,有利于模板DNA等規模化放大
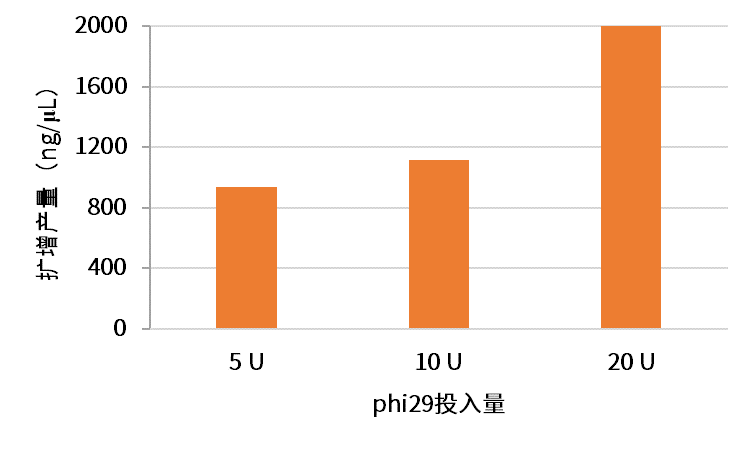
圖10. 翌圣phi29 DNA聚合酶擴增產量。模板:293細胞gDNA,投入100ng;反應時間:16h。結果顯示翌圣phi29 DNA聚合酶(Cat#14404ES)在100ng模板投入量下,擴增產量可達到2 μg/μL。
相關產品
應用分類 | 產品定位 | 產品名稱 | 貨號 |
酶法合成 | phi29 DNA 聚合酶 | phi29 DNA Polymerase (10 U/μL) | 14404ES |
TelN端粒酶 | TelN Protelomerase (5 U/μL) | 14540ES | |
核酸外切酶 | Exonuclease III | 14525ES | |
dNTP | dNTP Mix(25 mM each) | 10125ES | |
焦磷酸酶 | Inorganic Pyrophosphatase | 10658ES | |
單細胞測序 | 單細胞逆轉錄 | YeaCell™ 1st Strand Synthesis kit for Single Cell 3?RNA-seq | 13594ES |
單細胞/低投入量cDNA合成及擴增 | Hieff NGS® Single Cell/Low Input cDNA Synthesis & Amplification Module | 12500ES | |
單細胞RNA建庫 | Hieff NGS® Single Cell/Low Input RNA Library Prep Kit | 12502ES |
參考文獻
[1] Kamtekar S, Berman AJ, Wang J, et al. Insights into strand displacement and processivity from the crystal structure of the protein-primed DNA polymerase of bacteriophage phi29. Mol Cell. 2004;16(4):609-618.
[2] Rato S, Golumbeanu M, Telenti A, Ciuffi A. Exploring viral infection using single-cell sequencing. Virus Res. 2017;239:55-68.
[3] Huang L, Ma F, Chapman A, Lu S, Xie XS. Single-Cell Whole-Genome Amplification and Sequencing: Methodology and Applications. Annu Rev Genomics Hum Genet. 2015;16:79-102.
[4] Rato S, Golumbeanu M, Telenti A, Ciuffi A. Exploring viral infection using single-cell sequencing. Virus Res. 2017;239:55-68.

 初級會員·13年
初級會員·13年
