聯系電話
- 聯系人:
- 曹女士
- 電話:
- 400-6111-883
- 手機:
- 售后:
- 4006-111-883
- 傳真:
- 86-21-34615995
- 地址:
- 上海市浦東新區天雄路166弄1號3樓
- 網址:
- www.yeasen.com
掃一掃訪問手機商鋪
近年來,隨著生物醫藥迅猛發展以及基因細胞治療領域研究的深入,如何把控生物制品安全可靠,成為各國法規政府重視以及監管的要點。因為支原體是一種比較常見但通常難以去除的污染類型,所以對于涉及細胞培養的生物制品工藝過程,法規要求“都必須確保無支原體污染"。
監管機構要求的支原體檢測點
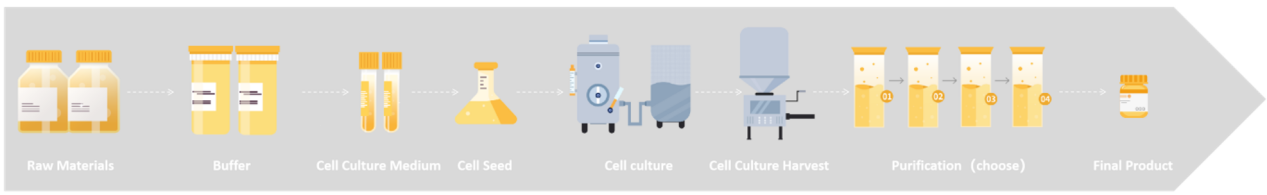
2020版中國藥典三部《生物制品生產檢定用動物細胞基質制備及質量控制》中提出對于生產用細胞,需要對主細胞庫(MCB)、工作細胞庫(WCB)、生產終末細胞(EOPC)進行支原體檢查;
《免疫細胞治療產品藥學研究與評價技術指導原則(試行)》中建議在關鍵時間點對適合的中間樣品開展支原體等安全性相關檢測或采取相關的措施加以控制,并且支原體同樣需要作為終產品的放行檢項;
FDA,Guidance for Industry :Characterization and Qualification of Cell Substrates and Other Biological Materials Used in the Production of Viral Vaccines for Infectious Disease Indications,中提出對于原材料、病毒種子、未加工的收獲液等都需要進行支原體控制。
快檢法和傳統方法的比較
在快速檢測方法(NAT,核酸擴增技術)出現之前,傳統的培養法和指示細胞法因其檢測周期長或靈敏度問題,致使企業生產或產品放行周期拉長或者需要基于風險評估對細胞原料進行條件放行或者尋求其他替代方法。而隨著細胞基因治療的發展,行業內對支原體檢測時效性和靈敏度要求越來越高,短的貨架期不足以支撐如此長的檢測周期,因此NAT方法從眾多支原體檢測方法中脫穎而出。
目前歐洲藥典(EP)<2.6.7>,日本藥典(JP)以及美國藥典(USP)<63>都已收錄NAT方法作為支原體檢測方法,但需要對該方法進行驗證以及與傳統方法進行對比其檢測靈敏度不低于傳統方法,才可以使用。2020版中國藥典(ChP)雖然并未收錄NAT法作為支原體檢測方法,但其中也提到了“也可采用經國家藥品檢定機構認可的其他方法"。2022年5月,國家藥監局藥審中心發布的《免疫細胞治療產品藥學研究與評價技術指導原則(試行)》中提到“當檢驗樣品量有限,或需要快速放行等特殊情況下,如藥典方法無法滿足,可考慮開發新型的無菌和支原體的檢測方法進行放行檢測,但是新型檢測方法應經過充分驗證"。所以NAT法有望作為能夠支持快速支原體放行檢測的方法,被越來越多的原材料供應商、以及ATMP企業所使用。
快檢法(NAT)-方法驗證要求
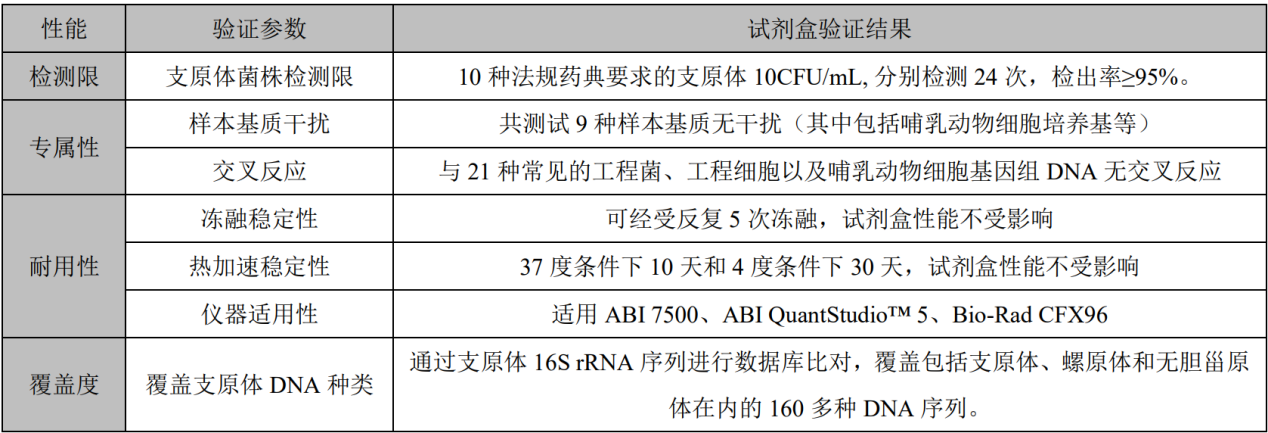
對于如何進行NAT方法的驗證,目前歐洲藥典<2.6.7>和日本藥典里都詳細介紹,其內容基本一致。中檢院也發表了文章《支原體檢查的核酸檢測方法及方法學驗證的思考》,以期對我國支原體核酸擴增法檢測的研發者及使用者提供借鑒。支原體NAT法須進行專屬性(Specificity)、檢測限(Detection Limit)和耐用性(Robustness)驗證。如果使用商業化試劑盒進行支原體檢測,則可以基于供應商的完整試劑盒性能驗證報告再結合本身實驗室環境和樣品類型進行合適的方法適用性驗證。
專屬性(Specificity)
專屬性系指在其他成分(如雜質、降解產物、輔料等)可能存在下,采用的分析方法能正確測定出被測物的能力。EP中提到需要在驗證中關注與系統發育關系比較近的其他細菌種屬之間的交叉反應,如革蘭氏陽性菌包括梭菌屬Clostridium、乳桿菌屬Lactobacillus和鏈球菌屬Streptococcus。當然也需要關注宿主細胞和實驗操作環境中比較常見的如人源DNA污染類型。
檢測限(Detection Limit)
檢測限系指試樣中被測物能被檢測出的低量。檢測限僅作為限度試驗指標和定性鑒別的依據,不需要被準確定量為一個準確值。EP中要求每種支原體每個稀釋濃度都需要有24次檢測數據,可以在不同天做3次獨立的10倍系列梯度稀釋,每個稀釋梯度做8次重復檢測;或者可以選擇不同天做4次獨立的10倍系列梯度稀釋,每個稀釋梯度做6次重復檢測。檢測陽性率需要滿足95%以上可以作為檢測限。
對需要進行檢測限確認的支原體類型,試劑盒廠家需盡可能多的覆蓋法規藥典中要求的支原體類型,并需要對每種支原體的檢測限進行摸索。而對于生物制品企業來說,通常需要根據實際使用的原輔料來源進行選擇。另外建議把人源支原體類型考慮進去。
耐用性系指在測定條件有小的變動時,測定結果不受影響的承受程度,為所建立的方法用于常規檢驗提供依據。對于耐用性的評估,需要關注支原體檢測方法對于檢測試劑中MgCl2、引物和dNTP的濃度變化,核酸提取試劑盒或提取步驟的改變以及使用不同核酸擴增儀的耐受程度。
翌圣生物商業化支原體快檢試劑盒(NAT,qPCR法)
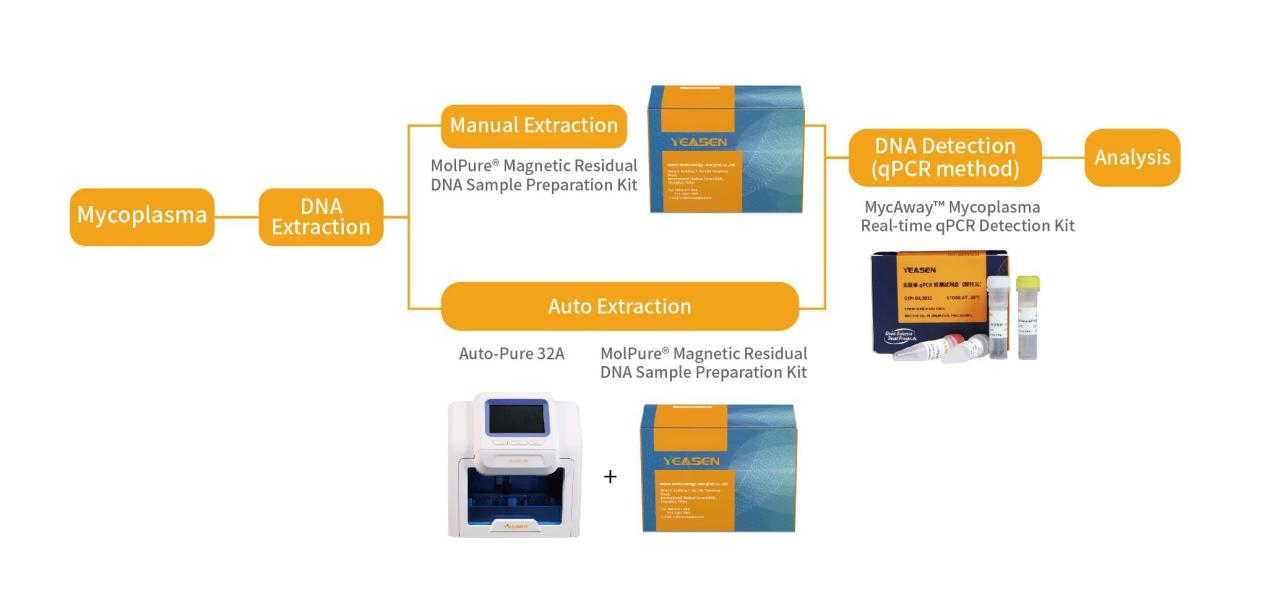
MycAway™支原體qPCR檢測試劑盒(探針法)是基于NAT的一種快速定性檢測生產原料、細胞庫、病毒種子、病毒或細胞收獲液、治療用細胞中潛在支原體污染的產品。該試劑盒基于定量PCR技術,使用Taqman熒光探針定性檢測待測樣本中支原體DNA,可覆蓋160多種支原體DNA序列;并且嚴格按照EP 2.6.7和JP G3支原體檢測相關指南和要求進行驗證,具備靈敏度高、特異性好、安全性好等特點。
它能夠與MolPure® 磁珠法殘留DNA樣本前處理試劑盒搭配使用,可以通過手動提取或者使用Auto-Pure 32A全自動核酸提取儀自動提取樣本核酸。樣本經過前處理去除干擾雜質獲得純化的支原體DNA后,通過qPCR收集探針的熒光信號,從而對檢測結果進行判定。

翌圣生物支原體qPCR檢測服務(樣品適用性驗證)
根據EP 2.6.7和JP XVII G3中的要求,用戶在使用商業化試劑盒進行支原體檢測時,除了可以使用試劑盒供應商提供的試劑盒性能驗證數據之外,還需要根據本身實驗環境和條件并針對自己的樣品,進行支原體檢測方法的方法適用性驗證。驗證參數包括:檢測限、專屬性、耐用性以及對比實驗。
翌圣生物針對質控類檢測試劑建立了相應的研發和生產平臺。其中,研發平臺以雙向分子酶設計與定向進化平臺和蛋白高密度發酵與超潔凈純化平臺為依托,建立分子診斷試劑關鍵原料研發平臺和獨立的分子和免疫檢測產品開發實驗室,并配備了先進的研發儀器設備。生產平臺,嚴格按照ISO13485質量體系標準建設和管理,擁有獨立的潔凈生產車間,滿足不同產品定量和定性的要求。層層把關,每一步的放行,都經過多項嚴格而高標準的質量驗收。
基于上述研發和生產平臺,翌圣生物可針對生物制品研發和生產過程中的樣本,提供配套翌圣生物“支原體qPCR檢測試劑盒(探針法)"(貨號:40618ES)的方法適用性驗證服務。

(最終解釋權歸翌圣生物所有)
參考文獻:
1.支原體檢查法<3301>.《中國藥典》三部[S]. 北京:中國科學技術出版社,2020:542-543.
2.分析方法驗證指導原則<9101>.《中國藥典》三部[S]. 北京:中國科學技術出版社,2020:682.
3.藥品微生物檢驗替代方法驗證指導原則<9201>.《中國藥典》三部[S]. 北京:中國科學技術出版社,2020:684.
4.Mycoplasmas. European Pharmacopoeia[S]. 10th Edition, 2020: Section 2.6.7, 194-199.
5.Mycoplasma Testing for Cell Substrates used for the Production of Biotechnological/Biological Products <G3-14-170>. The Japanese Pharmacopoeia[S]. 18th Edition, 2021: 2678-2682.
6.Erica J. Fratz-Berilla, Phillip Angart, Ryan J.Graham, et al. Impacts on product quality attributes of monoclonal antibodies produced in CHO cell bioreactor cultures during intentional mycoplasma contamination events. Biotechnology and Bioengineering. 2020;117(9): 2802-2815.
7.Dmitriy V. Volokhov, Laurie J Graham, Kurt A Brorson, Vladimir E Chizhikov. Mycoplasma testing of cell substrates and biologics: Review of alternative non-microbiological techniques. Mol Cell Probes. 2011 Apr-Jun;25(2-3):69-77.
8.A.L. Ingebritson, C.P. Gibbs, C. Tong, G.B. Srinivas. A PCR detection method for testing Mycoplasma contamination of veterinary vaccines and biological products. Lett in Applied Microbiology. 2015 Feb;60(2):174-180.

 初級會員·13年
初級會員·13年
